Клеточные и днк процессоры это
Обновлено: 07.07.2024
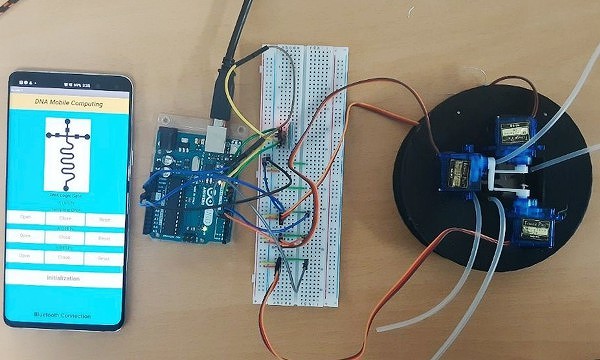
Группа ученых из Национального университета в Инчхоне (INU), Южная Корея, объявила о разработке первой в мире технологии выполнения классических вычислений на базе молекул ДНК. Свое изобретение исследователи назвали «Микрожидкостный процессор» (Microfluidic Processing Unit, MPU).
Разработка южнокорейских ученых впервые описана в статье Programmable DNA-Based Boolean Logic Microfluidic Processing Unit («Программируемый микрожидкостный процессор для работы с двоичной логикой на основе ДНК), которая была опубликована в последнем выпуске научного журнала ACS Nano, выпускаемого Американским химическим обществом (American Chemical Society). В ней подробно описан лабораторный прототип MPU, который выполнен в компактном форм-факторе, включает элементы обработки ДНК для исполнения ряда основных операций двоичной (булевой) логики, и может быть запрограммирован с обычного ПК или смартфона.
По словам ученых, уже первый прототип MPU смог выполнять ключевые операции AND, OR, XOR и NOT, работа с которыми подтвердила возможность использования молекул ДНК не только для хранения данных – как считалось ранее, но и для полноценных вычислений.
«Мы надеемся, что в будущем процессоры на основе ДНК заменят традиционные электронные чипы, поскольку они потребляют меньше энергии, и это поможет с глобальным потеплением, сказал – руководитель исследования из INU д-р Ёнджун Сонг (Dr. Youngjun Song). – Процессоры на базе ДНК также позволят создать платформы для сложных вычислений – таких как задачи глубокого машинного обучения и математического моделирования».
Как сделать свой ДНК-процессор
Ранее использование молекул ДНК для вычислительных нужд обсуждалось в научной среде преимущественно в ключе использования таких решений для хранения информации. Молекулы ДНК действительно подходят для решений, способных хранить огромные объемы данных, однако минусом такой технологии является чрезвычайно низкая скорость чтения и записи – до одной секунды на запись одной базы данных средних размеров в хранилище на основе ДНК.
Проблема в первую очередь связана с принципом хранения данных в ДНК, который требует другого подхода к обработке информации. По мнению ученых, естественным решением проблемы стал бы процессор на молекулах ДНК с аналогичным принципом форматирования данных. Именно в поисках решения этого вопроса работали южнокорейские ученые.
Как пояснили в своей статье исследователи, использование закодированных информацией цепочек дезоксирибонуклеиновой кислоты (ДНК) в качестве материала для молекулярных вычислений обеспечивает логический вычислительный процесс за счет каскадных и параллельных цепных реакций, однако реакции при вычислениях на основе комбинационной логики на основе ДНК в основном достигаются посредством ручного процесса путем добавления желаемых молекул ДНК в одну микропробирку (субстрат).
Как показало исследование, использование микрожидкостных микросхем на основе ДНК для операций булевой логики (логика «истинно/ложно», где сравниваются входящие данные и возвращается значение «истина» или «ложь» в зависимости от типа операции или используемого «логического элемента» - прим. CNews) может обеспечить автоматизированную работу, программируемое управление и бесшовную комбинационную работу логики по аналогии с электронным микропроцессором.
Для изготовления микрожидкостного молекулярного ДНК-чипа, способного выполнять булеву логику, команда ученых использовала систему 3D-печати с использованием методов двустороннего формования. Логический вентиль в лабораторном эксперименте состоял из одноцепочечной ДНК-матрицы, а в качестве входных данных использовались различные одноцепочечные ДНК.
«ВТБ Лизинг» внедряет управление данными как ценным бизнес-активом
Когда часть входной ДНК имела последовательность Уотсона-Крика, комплементарную матричной ДНК, она спаривалась и образовывала двухцепочечную ДНК, при этом результат считался истинным или ложным в зависимости от размера окончательной ДНК.
Управление прототипом ДНК-процессора осуществлялось необычным способом – с помощью системы клапанов с электроприводом, которая выполняет серию реакций для быстрого и удобного исполнения комбинации логических операций. Эту систему клапанов можно программировать с помощью ПК или смартфона.
Совокупность ДНК-чипа и управляющего ПО в итоге и была названа «микрожидкостным процессором» (MPU).
Дальнейшие перспективы проекта
По мнению авторов проекта, разработанная ими система клапанов для программируемого MPU на основе ДНК открывает путь к созданию более сложных систем с каскадами реакций, которые смогут кодировать расширенный список функции.
«Будущие исследования будут сосредоточены на создании вычислительных решений полностью на основе ДНК с алгоритмами ДНК и системами хранения ДНК», - говорит д-р Сонг.
Пока что для управления чипом необходима внешняя система, однако ученые полагают, что в перспективе удастся сформировать MPU булевой логики на основе ДНК, которым можно будет программировать с помощью специального языка программирования. Такие системы, по мнению разработчиков, смогут найти широкое распространение в более сложных функциональных решениях – таких как модули арифметических вычислений или нейроморфные схемы.

С учетом того, что закон Мура является всего лишь эмпирическим наблюдением и упирается в физическую вместимость микропроцессора, то есть, в количество транзисторов, которые можно уместить на единицу площади, вполне логично, что программно-аппаратная инженерия пытается уйти от традиционных носителей информации на материале соединений кремния. Тем более, что срок действия закона Мура явственно подходит к концу. Возможной альтернативой для вычислительной неорганики много лет мыслится вычислительная органика. То есть, теоретически, а также (возможно) практически должны быть варианты хранения информации в белках и нуклеиновых кислотах. Тем более, что нуклеиновые кислоты в природе превосходно справляются с кодированием и передачей информации.
Сразу оговоримся, что для информации нужно не только хранилище; нужен еще и процессор, а также устройства ввода-вывода. Поскольку до создания подобной инфраструктуры еще очень далеко, тема казалась бы спекулятивной, но в январе 2021 года в журнале «Nature of Chemical Biology» была опубликована статья, описывающая довольно простую технологию кодирования 3-битных информационных последовательностей в ДНК. Вот о чем она.
Двойная спираль. Винтовая лестница в обход закона Мура
В современном мире постоянно генерируется все больше данных, и исследователи как могут изобретают новые способы их хранения. ДНК по-прежнему считается весьма перспективной в качестве исключительно компактного и устойчивого носителя информации. А прямо сейчас формируется новый подход, позволяющий записывать цифровые данные непосредственно в геномы живых клеток.
Попытки переориентировать технологии запоминания данных, изобретенные природой, не новы, но в последнее десятилетие интерес к таким подходам оживился, и уже есть заметные достижения в этой области. Ситуация вызвана взрывным ростом генерируемых данных, причем, нет никаких признаков его замедления. Предполагается, что в 2025 году во всем мире ежедневно будет создаваться 463 эксабайт данных.
Хранение всех этих данных с применением кремниевых технологий вскоре может стать непрактичным, но выход может заключаться в использовании ДНК. Во-первых, плотность информации ДНК в миллионы раз выше, чем на обычных жестких дисках. Всего в одном грамме ДНК можно хранить до 215 миллионов гигабайт данных.
Кроме того, при правильном хранении ДНК исключительно стабильна. В 2017 году ученым удалось полностью восстановить геном лошади (вымершего вида), жившей 700 000 лет назад. Научившись хранить данные и обращаться с ними на том же языке, который используется в природе, мы открываем путь к множеству новых биотехнологических возможностей.
Основная сложность заключается в том, чтобы найти интерфейс между цифровым миром информатики и биохимическим миром генетики. В настоящее время для этого требуется синтезировать ДНК в лаборатории, и этот процесс по-прежнему дорогой и сложный, хотя, стоимость синтеза ДНК быстро снижается. Полученные последовательности затем тщательно хранятся in vitro, пока не потребуется вновь к ним обратиться, либо их можно внедрять в живые клетки при помощи технологии CRISPR, предназначенной для редактирования генов.
CRISPR – это популярная новая технология редактирования геномов, именно за нее была вручена Нобелевская премия по химии в 2020 году. Аббревиатура CRISPR означает «короткие палиндромные повторы, регулярно расположенные группами». Подробнее о ней можно почитать в замечательной свежей статье на Хабре. Здесь же оговоримся, что в интересующем нас контексте криспры могут использоваться так: бактерий стимулируют электрическим сигналом, заставляя таким образом вставлять в ДНК заранее определенные последовательности, соответствующие нулям и единицам. Статья об этом была опубликована 24 января 2021 года. Отметим, что применение электрических сигналов для встраивания криспров – это инновационный метод, ранее применялись только биохимические взаимодействия, например, индуцируемые фруктозой. Кроме того, хранение информации в клетках кишечной палочки исключительно эффективно – согласно более раннему источнику, в клетках кишечной палочки можно зашифровать 10 19 бит информации на кубический сантиметр. При дальнейшей экстраполяции можно вычислить, что ДНК, необходимую для хранения всех данных, имевшихся в распоряжении человека на 2017 год, можно уложить в виде куба с гранью 1 м.
Остановимся подробнее на описании этого эксперимента.
Авторы заменили систему ввода, использующую индуцирование фруктозой на систему электронного ввода, которая позволяет кодировать более длинные последовательности. При помощи синтетической адаптивной системы CRISPR, работающей по принципу редокс-отклика, удалось усилить экспрессию генов в ответ на рост электрического напряжения. Таким образом цифровые данные были закодированы непосредственно в бактерии, без необходимости синтеза ДНК in vitro.

Итак, бактериальный биоматериал и молекула ДНК в частности удобны для хранения любой информации, а не только биологической. Кроме того, размножение бактерий решает проблему с резервным копированием данных, а информационная плотность лабораторной бактерии E.Coli более чем достаточна с учетом потребностей современной информатики.
Мембранные вычисления
Ниже в этой статье мы также затронем тему переноса логических операций из информатики на биоматериал, но до этого поговорим о концептуальном сходстве программирования и биологии, выраженном в концепции «мембранных вычислений». Здесь предлагается введение в мембранные вычисления (презентация). Концепция мембранных вычислений, впервые сформулированная в 1998 году, предполагает, что выстраивание программных систем и алгоритмов возможно по такому же восходящему принципу, что и формирование организма из отдельных клеток и тканей. Мембранные вычисления предполагают операции над группами (мультимножествами) объектов, каждое из которых логически находится в пределах мембраны, напоминающей клеточную, и может обмениваться данными с другими подобными мультимножествами. Мультимножества могут группироваться в совокупности, напоминающие биологические ткани, органы, либо колонии бактерий. При этом подобные структуры удобны для представления «клеток» (мультимножеств) в качестве узлов графа, а также хорошо поддаются распределенным и параллельным вычислениям. Возможные области применения парадигмы – биология, биомедицина, лингвистика, компьютерная графика, экономика, оптимизация, криптография.
Наряду с «клеточными» и «обычными тканевыми» мембранными системами наиболее интересны импульсные нейронные P-системы (spiking neural P-systems), которые сближаются по структуре с нервной тканью. В вышеприведенной презентации они рассматриваются на слайде 93 и далее и к 2020 году уже находят реальное практическое применение: например, в распознавании образов. Учитывая, что мембранные системы располагают к разработке специализированных эволюционных алгоритмов, в частности, для целей оптимизации, можно говорить о целом классе наработок, которые естественным образом подходят для реализации на молекулярно-биологическом материале.
Масштабирование вычислений и другие прикладные проблемы
Кроме того, принципиальное значение для традиционной вычислительной техники играют:
В статье 2017 года, подготовленной в MIT (Массачусетском технологическом институте) рассказано, как эти концепции могут быть реализованы на уровне биохимии. Память является фундаментальным элементом вычислительных систем, так как позволяет учитывать не только историю произошедших событий, но и события, происходящие в системе и ее окружении прямо сейчас. Масштабирование позволяет справляться с распределением вычислений, причем, в рассматриваемой области достигается простым увеличением числа клеток. Логику операций в ДНК можно закладывать на уровне сайтов присоединения, на месте которых ферменты-рекомбиназы могут вставлять, вырезать или переворачивать фрагменты ДНК, в зависимости от запрограммированной операции. Также при помощи ферментов можно программировать в клетке поведение, аналогичное работе машины состояний. Еще в 2013 году было показано, что при помощи ферментов в ДНК можно реализовать все логические операции, не создавая при этом каскадов логических вентилей. При этом отдельно придется программировать порядок действия ферментов, которые будут воздействовать на ДНК. Существуют исследования, описывающие разработку биологических машин состояний на основе рекомбинации ДНК. Для ввода и вывода в таких системах используются искусственно сформированные плазмиды, при внедрении которых в клетку эта клетка должна перейти в заданное состояние. Точность воспроизведения заданного состояния даже через три поколения близка к 100%.

Что касается возможных проблем такого подхода в продакшене – зависимость скорости операций, например, считывания данных, от скорости транскрипции белка. Также процесс передачи информации в живой клетке невозможно полностью защитить от мутаций, а скорость накопления мутаций, по-видимому, должна повышаться с ускорением работы таких эволюционных механизмов.
Интересно, что работа по проектированию логических вентилей на уровне белков проводится и для решения чисто биологических задач, например, для борьбы с истощением запаса иммунных Т-клеток. Авторы этой работы подчеркивают, что белки в качестве логических единиц могут работать и без клеточного аппарата, непосредственно в межклеточной среде, а синтез белков с заданными свойствами позволяет управлять и топологией их молекул, и принципами связывания с другими молекулами. Кроме того, белки несопоставимо более разнообразны по составу и химическим свойствам, нежели нуклеиновые кислоты, поэтому, по мнению авторов статьи, на уровне белков можно запрограммировать гораздо больше функциональных возможностей, чем при подходе, предполагающем манипуляции с CRISPR. Возможно, использование белков в таком качестве позволит создавать даже совершенно новые клетки и ткани с заданными свойствами. Но белки, тем не менее, принципиально проигрывают ДНК по показателям прочности, репликации и долговечности, поэтому целесообразно их использовать для программирования клеток – например, как раз такой искусственной ткани или синтетических бактерий, которые предназначались бы именно для хранения и обработки информации.
Клеточный процессор
Наиболее сложной готовой разработкой в этой области представляется настоящий двухъядерный клеточный процессор, собранный в Высшей Технической школе Цюриха (на факультете биотехнологии и биоинженерии, расположенном в Базеле) в 2019 году. Этот процессор, работающий на основе технологии CRISPR, получен путем внедрения в живую клетку второго клеточного ядра. В качестве ввода он принимает молекулы РНК, регулируя экспрессию заданных генов и управляя таким образом синтезом белков. Исследователи подчеркивают, что в качестве такого процессора может работать единственная двухъядерная клетка, но ничто не мешает масштабировать его до целой ткани, содержащей миллиарды двухъядерных клеток. Теоретически такая система может превзойти по мощности современный суперкомпьютер, но потреблять лишь малую толику энергии по сравнению с таким суперкомпьютером. К тому же, на практике ничто не мешает внедрить в клетку не два, а сколько угодно ядер.
Клеточный компьютер может использоваться для детектирования биомаркеров в качестве ввода и для соответствующего реагирования на этот ввод. В качестве вывода клеточный компьютер может синтезировать молекулу лекарства, либо диагностического вещества. Подобное устройство может применяться, например, при лечении рака.
Здесь также нельзя обойтись без упоминания потрясающих возможностей биохимических компьютеров в областях, сближающихся с 3D-печатью, конструированием мемристоров и решением задач на поиск кратчайшего пути. На Хабре есть интересная статья «Нейросеть с амебой решили задачу коммивояжера», но амебы не сравнятся в поиске пути со слизевиками, которые способны захватывать жизненное пространство, вырастая от
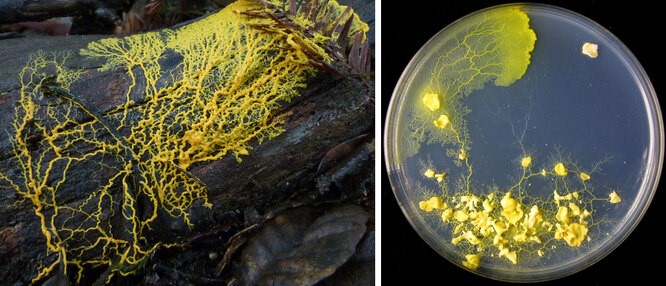

не обладая никакой нервной системой. Фактически, эти организмы состоят из сплошной цитоплазмы и захватывают окружающее пространство, достраивая себя в соответствии с некими топологическими алгоритмами. Например, слизевик успешно «запоминает», как прорасти к источнику пищи, даже если после достижения пищи отсечь его отросток. Программная реализация таких способностей позволила бы моделировать и создавать материалы с заданными свойствами, поскольку могла бы работать на клеточном и даже молекулярном уровне.
Здесь весьма кратко обрисованы возможности и проблемы биотехнологического программирования, но тема представляется очень перспективной во многом потому, что одни и те же законы клеточной репликации позволяют работать и с нуклеиновыми кислотами, и с белками/ферментами. Освоение подобных возможностей позволило бы сочетать долговечность и информационную избыточность ДНК (хранилище данных) с разнообразием белков (структуры данных), и не только кардинально решить проблему исчерпания возможностей закона Мура, но и полностью переосмыслить вычислительную технику, заменив software+hardware на wetware.
В настоящее время в поисках реальной альтернативы полупроводниковым технологиям создания новых вычислительных систем ученые обращают все большее внимание на биотехнологии, или биокомпьютинг , который представляет собой гибрид информационных, молекулярных технологий, а также биохимии. Биокомпьютинг позволяет решать сложные вычислительные задачи, используя методы, принятые в биохимии и молекулярной биологии, организуя вычисления при помощи живых тканей, клеток, вирусов и биомолекул. Наибольшее распространение получил подход, где в качестве основного элемента (процессора) используются молекулы дезоксирибонуклеиновой кислоты . Центральное место в этом подходе занимает так называемый ДНК-процессор . Кроме ДНК, в качестве биопроцессора могут использоваться также белковые молекулы и биологические мембраны .
ДНК-процессоры
Так же, как и любой другой процессор, ДНК-процессор характеризуется структурой и набором команд. В нашем случае структура процессора – это структура молекулы ДНК. А набор команд – это перечень биохимических операций с молекулами. Принцип устройства компьютерной ДНК-памяти основан на последовательном соединении четырех нуклеотидов (основных кирпичиков ДНК-цепи). Три нуклеотида, соединяясь в любой последовательности, образуют элементарную ячейку памяти – кодон, совокупность которых формирует затем цепь ДНК. Основная трудность в разработке ДНК-компьютеров связана с проведением избирательных однокодонных реакций (взаимодействий) внутри цепи ДНК. Однако прогресс есть уже и в этом направлении. Существует экспериментальное оборудование, позволяющее работать с одним из 1020 кодонов или молекул ДНК. Другой проблемой является самосборка ДНК, приводящая к потере информации. Ее преодолевают введением в клетку специальных ингибиторов – веществ, предотвращающих химическую реакцию самосшивки.
Использование молекул ДНК для организации вычислений – это не слишком новая идея. Теоретическое обоснование подобной возможности было сделано еще в 50-х годах прошлого века (Р.П. Фейманом). В деталях эта теория была проработана в 70-х годах Ч. Бенеттом и в 80-х М. Конрадом.
Первый компьютер на базе ДНК был создан еще в 1994 г. американским ученым Леонардом Адлеманом. Он смешал в пробирке молекулу ДНК, в которой были закодированы исходные данные, и специальным образом подобранные ферменты. В результате химической реакции структура ДНК изменилась таким образом, что в ней в закодированном виде был представлен ответ задачи. Поскольку вычисления проводились в ходе химической реакции с участием ферментов, на них было затрачено очень мало времени.
Вслед за работой Адлемана последовали другие. Ллойд Смит из Университета Висконсин решил с помощью ДНК задачу доставки четырех сортов пиццы по четырем адресам, которая подразумевала 16 вариантов ответа. Ученые из Принстонского университета решили комбинаторную шахматную задачу: при помощи РНК нашли правильный ход шахматного коня на доске из девяти клеток (всего их 512 вариантов).
Ричард Липтон из Принстона первым показал, как, используя ДНК, кодировать двоичные числа и решать проблему удовлетворения логического выражения. Суть ее в том, что, имея некоторое логическое выражение, включающее n логических переменных, нужно найти все комбинации значений переменных, делающих выражение истинным. Задачу можно решить только перебором 2n комбинаций. Все эти комбинации легко закодировать с помощью ДНК, а дальше действовать по методике Адлемана. Липтон предложил также способ взлома шифра DES (американский криптографический), трактуемого как своеобразное логическое выражение.
Первую модель биокомпьютера, правда, в виде механизма из пластмассы, в 1999 г. создал Ихуд Шапиро из Вейцмановского института естественных наук. Она имитировала работу "молекулярной машины" в живой клетке, собирающей белковые молекулы по информации с ДНК, используя РНК в качестве посредника между ДНК и белком.
А в 2001 г. Шапиро удалось реализовать вычислительное устройство на основе ДНК, которое может работать почти без вмешательства человека. Система имитирует машину Тьюринга — одну из фундаментальных концепций вычислительной техники. Машина Тьюринга шаг за шагом считывает данные и в зависимости от их значений принимает решения о дальнейших действиях. Теоретически она может решить любую вычислительную задачу. По своей природе молекулы ДНК работают аналогичным образом, распадаясь и рекомбинируясь в соответствии с информацией, закодированной в цепочках химических соединений.
Разработанная в Вейцмановском институте установка кодирует входные данные и программы в состоящих из двух цепей молекулах ДНК и смешивает их с двумя ферментами. Молекулы фермента выполняли роль аппаратного, а молекулы ДНК – программного обеспечения. Один фермент расщепляет молекулу ДНК с входными данными на отрезки разной длины в зависимости от содержащегося в ней кода. А другой рекомбинирует эти отрезки в соответствии с их кодом и кодом молекулы ДНК с программой. Процесс продолжается вдоль входной цепи, и, когда доходит до конца, получается выходная молекула, соответствующая конечному состоянию системы.
Этот механизм может использоваться для решения самых разных задач. Хотя на уровне отдельных молекул обработка ДНК происходит медленно, со скоростью от 500 до 1000 бит/с, что во много миллионов раз медленнее современных кремниевых процессоров, по своей природе она допускает массовый параллелизм. По оценкам Шапиро и его коллег, в одной пробирке может одновременно происходить триллион процессов, так что при потребляемой мощности в единицы нановатт может выполняться миллиард операций в секунду.
Компьютер, построенный Olympus Optical, имеет молекулярную и электронную составляющие. Первая осуществляет химические реакции между молекулами ДНК, обеспечивает поиск и выделение результата вычислений. Вторая – обрабатывает информацию и анализирует полученные результаты.
Возможностями биокомпьютеров заинтересовались и военные. Американское агентство по исследованиям в области обороны DARPA выполняет проект, получивший название Bio-Comp (Biological Computations, биологические вычисления). Его цель – создание мощных вычислительных систем на основе ДНК.
Пока до практического применения компьютеров на базе ДНК еще очень далеко. Однако в будущем их смогут использовать не только для вычислений, но и как своеобразные нанофабрики лекарств. Поместив подобное "устройство" в клетку, врачи смогут влиять на ее состояние, исцеляя человека от самых опасных недугов.
Клеточные компьютеры представляют собой самоорганизующиеся колонии различных "умных" микроорганизмов, в геном которых удалось включить некую логическую схему, которая могла бы активизироваться в присутствии определенного вещества. Для этой цели идеально подошли бы бактерии, стакан с которыми и представлял бы собой компьютер. Такие компьютеры очень дешевы в производстве. Им не нужна стерильная атмосфера, как при производстве полупроводников.
Главное свойство такого компьютера состоит в том, что каждая его клетка представляет собой миниатюрную химическую лабораторию. Если биоорганизм запрограммирован, то он просто производит нужные вещества. Достаточно вырастить одну клетку, обладающую заданными качествами, и можно легко и быстро вырастить тысячи клеток с такой же программой.
Основная проблема, с которой сталкиваются создатели клеточных биокомпьютеров, – организация всех клеток в единую работающую систему. На сегодня практические достижения в области клеточных компьютеров напоминают достижения 20-х годов в области ламповых и полупроводниковых компьютеров. Сейчас в Лаборатории искусственного интеллекта Массачусетского технологического университета создана клетка, способная хранить на генетическом уровне 1 бит информации. Также разрабатываются технологии, позволяющие единичной бактерии отыскивать своих соседей, образовывать с ними упорядоченную структуру и осуществлять массив параллельных операций.
В 2001 г. американские ученые создали трансгенные микроорганизмы (т. е. микроорганизмы с искусственно измененными генами), клетки которых могут выполнять логические операции И и ИЛИ.
Специалисты лаборатории Оук-Ридж, штат Теннесси, использовали способность генов синтезировать тот или иной белок под воздействием определенной группы химических раздражителей. Ученые изменили генетический код бактерий Pseudomonas putida таким образом, что их клетки обрели способность выполнять простые логические операции. Например, при выполнении операции И в клетку подаются два вещества (по сути – входные операнды), под влиянием которых ген вырабатывает определенный белок. Теперь ученые пытаются создать на базе этих клеток более сложные логические элементы, а также подумывают о возможности создания клетки, выполняющей параллельно несколько логических операций.
Потенциал биокомпьютеров очень велик. К достоинствам, выгодно отличающим их от компьютеров, основанных на кремниевых технологиях, относятся:
- более простая технология изготовления, не требующая для своей реализации столь жестких условий, как при производстве полупроводников;
- использование не бинарного, а тернарного кода (информация кодируется тройками нуклеотидов), что позволит за меньшее количество шагов перебрать большее число вариантов при анализе сложных систем;
- потенциально исключительно высокая производительность, которая может составлять до 10 14 операций в секунду за счет одновременного вступления в реакцию триллионов молекул ДНК;
- возможность хранить данные с плотностью, в триллионы раз превышающей показатели оптических дисков;
- исключительно низкое энергопотребление.
Однако, наряду с очевидными достоинствами, биокомпьютеры имеют и существенные недостатки, такие как:
- сложность со считыванием результатов – современные способы определения кодирующей последовательности несовершенны, сложны, трудоемки и дороги;
- низкая точность вычислений, связанная с возникновением мутаций, прилипанием молекул к стенкам сосудов и т.д.;
- невозможность длительного хранения результатов вычислений в связи с распадом ДНК в течение времени.
Хотя до практического использования биокомпьютеров еще очень далеко, и они вряд ли будут рассчитаны на широкие массы пользователей, предполагается, что они найдут достойное применение в медицине и фармакологии, а также с их помощью станет возможным объединение информационных и биотехнологий.
Чтобы понять, что такое ДНК, сначала нужно разобраться в её структуре, понять из чего состоит эта макромолекула и какие взаимодействия в ней происходят. Сама аббревиатура ДНК расшифровывается, как дезоксирибонуклеиновая кислота. Отсюда вывод, что эта молекула относится к нуклеиновым кислотам. К ним же принадлежит и РНК.

Структура
Все видели, как внешне выглядит ДНК. Её изображения нам известны из фильмов, интернета, учебников биологии. В основании имеются две нити, которые закручиваются в спираль. Спираль, как правило, закручена вправо. Почему именно спираль? Дело в том, что сами нити состоят из азотистых оснований, остатков фосфорной кислоты и дезоксирибозы (отсюда и первая буква «Д» в аббревиатуре). Эти азотистые основания: аденин, тимин, урацил и гуанин по своей природе гидрофобные. А так как нити соединяются между собой водородными связями, то такие связи крайне невыгодны для азотистых оснований. Спиралевидная формы ДНК делает возможным такое взаимодействие.
Функции
ДНК содержится главным образом в ядрах клеток и митохондриях. При делении ядра происходит и деление дезоксирибонуклеиновой кислоты. Вся информация о строении скелета, мышечных тканей, цвете глаз, волос, даже предрасположенности к определённым заболеваниям зашифрованы в генетическом коде. Процесс деления ДНК называется репликация. Но это не просто деление на две или более частей – это копирование. Причем полное и безоговорочное. Это и есть важнейшая функция этой таинственной молекулы.
Сохранить и передать всю информацию в следующее поколение без изменений задача не легкая. Иногда случаются сбои. Такие сбои называются мутациями. В процессе создания новой цепочки ДНК одно, два или несколько азотистых оснований могут замениться на другие. Например, тимин поменялся на аденин. В результате образуется новая структура, которая будет дальше передаваться уже только в измененном виде.

Возможности
Генетика открывает невероятные горизонты для человечества. Еще каких-то 40-50 лет назад мы не могли и догадываться о возможностях нашего генома. Генетические анализы вошли в нашу жизнь и успешно удовлетворяют наши потребности. Точность и бескомпромиссность генетики семимильными шагами несет эту науку по планете.
Наиболее популярные тесты, которые можно сделать практически в любой ДНК лаборатории:
- Установление отцовства и родства;
- Определение этнического происхождения;
- Выявление хромосомных мутаций;
- Обнаружение наследственных заболеваний;
- Узнать индивидуальные возможности организма в спорте.
Все это и еще многое другое выполняет ДНК центр «ДТЛ». Мы уже на протяжении шести лет специализируется на генетических тестах. Мы поможет подобрать анализ, собрать образцы и ответим на любые Ваши вопросы.
Читайте также:

